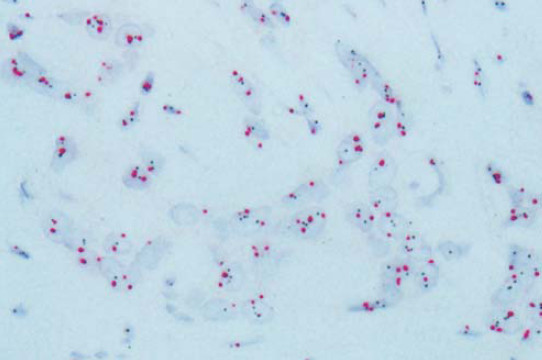
のシグナル、赤はセントロメア 17(CEP17)のシグナルである。参考までにHER2遺伝子は融合遺伝子ではなく、増幅されたHER2遺伝子のコピー数を測定する。HER2遺伝子が増えていると、この遺伝子のより産生される蛋白質(HER2蛋白)も増加しており、抗HER2療法による治療効果が期待される。ISH (in situ hybridization)とは、とは、組織中のRNAまたはDNAが存在する”原位置 (in situ) “をRNA-RNA、RNA-DNA、DNA-DNA ハイブリダイゼーションにより検出する方法である。組織切片を用いて特定遺伝子を検出することができ、特にがん、発生分化、形態形成の研究において有効な方法である。
融合遺伝子の検出について
日本血液学会 HP
最初に各種遺伝子異常の検出法について
遺伝子異常を検出可能な各検査法の特性を以下に示す。
1. 染色体検査:分裂中期の染色体をG-分染法等で染色、観察することで、染色体の異数性や構造異常(転座、逆位、欠失、増幅等)をゲノム全体にわたって、単一細胞レベルで評価することが可能な検査方法である。一般には、染色体検査のみで、異常に関わる標的遺伝子の特定に至ることは難しい。細胞の培養状態等により、分裂中期像が得られない場合には解析不能である。
2. FISH(fluorescent in situ hybridization):遺伝子座特異的な蛍光標識された核酸プローブを用いて、融合遺伝子の形成や遺伝子の再構成に至る染色体の構造異常やコピー数変化を検出する方法である。解析には間期核を用いるため、染色体検査で分裂中期像が得られない場合でも染色体の構造異常やコピー数変化を検出することが可能である。免疫グロブリン遺伝子座やT細胞受容体遺伝子座を含む染色体転座 (IGH-BCL2, IGH-MYCなど) や融合遺伝子を伴う逆位や転座(PML-RARA, NPM1-ALKなど)、また、遺伝子増幅を含むコピー数異常(MYC遺伝子の増幅など) の検出に有効である。
3. RT-qPCR (reverse transcription quantitative PCR) :リアルタイムPCR法とも呼ばれる。RNAから逆転写酵素を用いてcDNAを作製し、標的遺伝子特異的なPCRプライマーと蛍光プローブ、若しくは核酸と結合する蛍光色素を用いて標的RNAの発現量を定量解析する方法である。融合遺伝子の定量解析に有用であり、病型診断や測定可能残存病変・微小残存病変(MRD: measurable/minimal residual disease)の解析などに広く臨床応用されている。RNA-sequencingとは異なり、プライマーを設計した既知の融合遺伝子のみ検出可能である。
4. Target sequencing:一般的に「がん遺伝子パネル検査」に用いられる遺伝子異常の検出法である。相補的核酸プローブを用いたハイブリダイゼーション (targeted-capture sequencing) やPCRによるアンプリコン増幅 (amplicon sequencing) によって、標的とするゲノム領域を選択的に濃縮し、次世代シークエンサーを用いて網羅的に解析する。解析対象の遺伝子・遺伝子座が決まっている場合(がん遺伝子パネル検査など)には、シークエンス費用、必要データ量、解析に要する時間などの観点から、現状では 最も現実的なシークエンス法である。一般的に300×以上のシークエンス深度を得ることが可能であるため、腫瘍細胞のクローン構造解析やMRD解析にも応用可能である。また、RNAから逆転写された相補鎖DNAを用いたtargeted RNA-sequencingも可能であり、融合遺伝子の同定に有用である。
その他、以下のものがある。
MLPA(Multiplex Ligation-dependent Probe Amplification): 鋳型DNAにハイブリダイズしたプローブをPCRにより増幅することにより、対象ゲノム領域のコピー数変化を定量的に解析する方法である。ALLにおけるIKZF1など一部の遺伝子のコピー数減少の判定に用いられる場合がある。
WGS(whole genome sequencing):次世代シークエンサーを用いて、全ゲノム領域を解読する、最も包括的な手法である。1塩基変異(single-nucleotide variant: SNV)だけでなく、挿入・欠失(insertion or deletion: INDEL)、コピー数変化(copy-number variation: CNV、減少、増幅、ヘテロ接合性の消失などを含む)、大規模な構造異常(structural variation: SV、転座、逆位、欠失、重複などを含む)、外来性ゲノム(EBVやHTLV-1ゲノムなど)などを検出可能である。
WES(whole exome sequencing):次世代シークエンサーを用いて、ほぼすべての遺伝子のコーディングエクソン領域(タンパクをコードするゲノム領域)を解読する方法である。がんのドライバーとなる可能性の高い遺伝子領域を集中的に解読することで、解析時間と費用の効率化を実現できる。
Sanger sequencing:古典的なサンガーシークエンス法を用いた解析。対象とする遺伝子領域を特異的プライマーを用いてPCR増幅した後、増幅産物を解析する。スループットの観点から、解析可能な遺伝子数・領域には著しい制限があり、網羅的な遺伝子解析には適さない。異常の検出感度も低い。
ASO-PCR (allele-specific oligonucleotide PCR) :既知の遺伝子異常(ミスセンス変異など)の検出を高感度に行う方法である。既知の遺伝子異常に対応するプライマー、若しくはプライマー・プローブのセットを用いてPCRを行い、遺伝子異常を特異的に検出する方法である。MYD88 L265P変異、BRAF V600E変異などの検出に有効である。
RNA-sequencing:次世代シークエンサーを用いて、RNAから逆転写された相補鎖DNAを解読することにより、トランスクリプトーム全体を評価する方法である。遺伝子の発現定量 やスプライシングバリアントの検出、融合遺伝子の同定などに有用であり、未知の異常を含めて網羅的な探索が可能である。遺伝子異常の検出も可能であるが、変異の検出精度は低い。
k. CGH(comparative genomic hybridization)/array CGH/SNPsアレイ:ゲノムのコピー数変化を網羅的に解析する方法である。腫瘍組織と対照組織から抽出したゲノムDNAを異なる蛍光色素で標識をしたのちに混合して分裂中期の染色体にハイブリダイゼーションを行う。蛍光シグナルの比を観察することで、腫瘍組織のゲノムコピー数変化を検出する。array CGHは分裂中期の染色体の代わりに、数百~数千のDNAプローブをスライドグラス上に配置したものに対してハイブリダイゼーションを行うことで解像度を数十Kbに高めたものである。さらにSNPsアレイは数十万のSNPsタイピングを行うマイクロアレイをコピー数解析に応用したものでありアレル不均衡を検出することができる。


コメント